Creen la base de dades més extensa sobre proteïnes priòniques
Sorprenentment, són molts els organismes amb proteïnes priòniques al seu genoma. En la seva forma correcta, aquestes proteïnes intervenen en processos biològics fonamentals, com ara el desenvolupament del cervell o la diferenciació cel·lular en els humans. Però un plegament anormal en la seva estructura les converteix en prions, una versió agregada i nociva implicada en malalties neurodegeneratives com l’esclerosi lateral amiotròfica i la demència frontotemporal.
Aquestes proteïnes contenen regions amb seqüències específiques d’aminoàcids que en condicions fisiològiques normals els permeten formar assemblatges cel·lulars molt dinàmics, fluctuant entre formes solubles i agregades de manera natural. En humans, quan aquest equilibri es decanta cap a la forma agregada per mutacions genètiques o estrès, la proteïna es converteix en un prió i dóna lloc a malalties neurodegeneratives.
Encara queda, però, molt per descobrir sobre els mecanismes que les regulen i el seu funcionament. El nombre de proteïnes caracteritzades fins ara és molt baix, només n’hi ha un grapat en llevat i mamífers. Per a la seva identificació s’han emprat diversos models predictius, però tots han resultat ineficaços per l’alt nivell de falsos positius detectats.
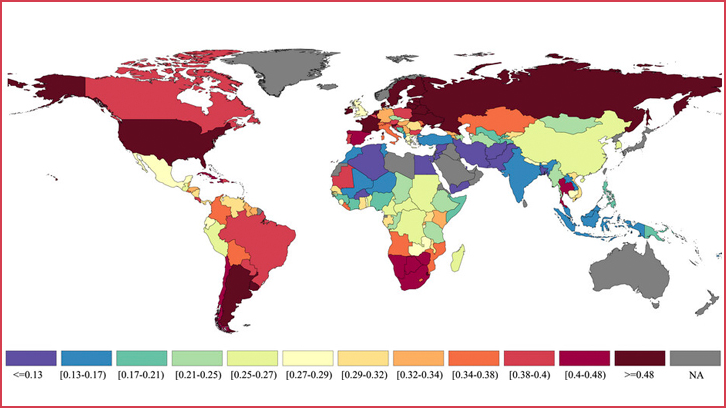
La base de dades PrionScan és fruit d’una nova estratègia computacional desenvolupada pels investigadors de la UAB i de la Universitat de Saragossa que explota les diferències seqüencials entre prions i no-prions i resulta molt més precisa que els models previs. El nou model ha permès identificar fins a 28.000 proteïnes susceptibles de transformar-se en prions en més de 3.200 organismes en tots els regnes de la vida, des de virus fins a plantes i mamífers.
“Hem trobat que la presència de proteïnes amb potencialitat per comportar-se com a prions és molt més gran del que havíem pensat. En organismes inferiors són especialment freqüents en els patogènics, com Plasmodium o Staphylococus. Dades preliminars suggereixen que el seu dinamisme ajudaria a aquests patògens a sobreviure a antibiòtics o a la resposta immunitària. En mamífers, i especialment en humans, estan implicades en processos molt importants com la formació del cervell i el desenvolupament embrionari. Així doncs, aquestes proteïnes fan funcions diferents en diferents organismes, però totes exploten la capacitat de fluctuar entre estats solubles i agregats”, explica Salvador Ventura, investigador del Departament de Bioquímica i Biologia Cel·lular i de l’Institut de Biotecnologia i Biomedicina de la UAB.
PrionScan s’actualitza regularment amb la totalitat de seqüències de proteïnes disponibles en bases de dades i pot ser utilitzat per fer cerques o analitzar seqüències de proteïnes introduïdes pels usuaris. Les cerques poden ser combinades per generar patrons de cerca més complexos i específics.
“Esperem que la nostra base de dades contribueixi a fer descobriments rellevants sobre les funcions i la regulació dels prions des d’una perspectiva genòmica. Hauria de permetre generar noves estratègies terapèutiques contra patògens que les continguin, identificar noves proteïnes implicades en malalties neurodegeneratives en humans i dissenyar nous prions sintètics, que podrien ser utilitzats com a interruptors moleculars en biologia sintètica”, conclou Ventura.
Referències
Espinosa Angarica, Vladimir; Ventura, Salvador; Sancho, Javier. Discovering putative prion sequences in complete proteomes using probabilistic representations of Q/N-rich domains. BMC Genomics 14: 316. 2013.
Espinosa Angarica, Vladimir; Angulo, Alfonso; Giner, Arturo; Losilla, Guillermo; Ventura, Salvador; Sancho, Javier. PrionScan: an online database of predicted prions domains in complete proteomes. BMC Genomics 15: 102. 2013.