eMIRNA: una nova eina informàtica per a detectar gens microRNA
La ramaderia porcina és el sector de major impacte econòmic a Catalunya. Per aquest motiu és fonamental aprofundir en la comprensió de la fisiologia d'aquest animal. Conjuntament des del Centre de Recerca en Agrigenòmica (CRAG) i la UAB, un grup d'investigadors ha desenvolupat una nova eina bioinformàtica, eMIRNA, que permet localitzar i identificar en porc i en qualsevol espècie els gens microRNA, petites molècules importants en la regulació de nombrosos caràcters productius d'interès. Aquesta eina ofereix, a més, l'oportunitat de millorar els catàlegs d'aquest tipus de gens que ja existeixen amb anterioritat.
Els microRNAs són petites molècules de RNA encarregades de regular l'expressió dels gens i adequar-la a les necessitats fisiològiques de l'organisme. En aquest sentit, els microRNAs tenen la capacitat d'unir-se a RNAs missatgers i propiciar la seva degradació o reprimir la seva traducció a proteïnes en els ribosomes. Així mateix, poden influir decisivament en l'activació o repressió de nombroses rutes metabòliques i regular processos biològics rellevants i molt variats. És per tant molt important localitzar i identificar els gens microRNA presents en el genoma d'una espècie amb l'objectiu de caracteritzar la seva seqüència i la seva funció biològica, així com detectar possibles mutacions que poguessin afectar la seva activitat reguladora.
Un dels principals obstacles que els investigadors troben a l'hora d'estudiar els gens microRNA és precisament la manca de catàlegs fiables en nombroses espècies de gran interès productiu, com són el porc, l'ovella o la cabra. Això demostra la imperiosa necessitat de disposar d'eines informàtiques que permetin identificar i caracteritzar aquest tipus de gens microRNA, a partir de qualsevol genoma animal, amb la finalitat de construir catàlegs el més complets possibles.
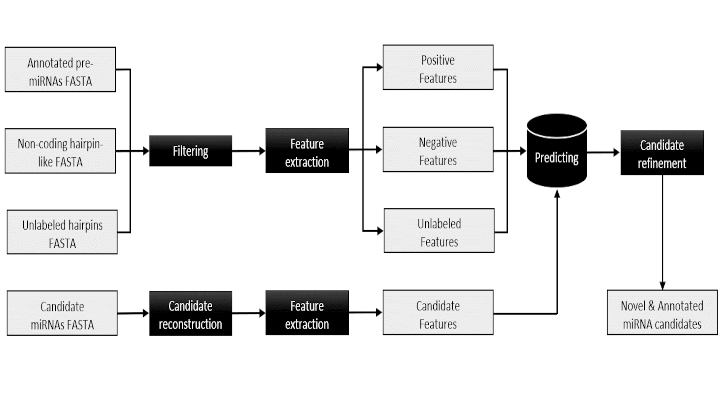
En aquest estudi, publicat a la revista Genomics, hem desenvolupat una eina bioinformàtica que permet la predicció i anotació funcional de microRNAs. Aquesta eina comprèn diferents mòduls descarregables de fàcil ús i adaptabilitat. Una descripció detallada d'aquesta metodologia es pot trobar a: https://github.com/emarmolsanchez/eMIRNA. La nostra motivació va partir de la necessitat d'aprofundir en l'anotació de microRNAs en el genoma porcí, una de les espècies domèstiques amb major impacte econòmic al sector ramader a Catalunya.
Una de les principals innovacions de l'eina eMIRNA va consistir a demostrar l'eficàcia d'incloure la recerca de motius nucleotídics en la reconstrucció de gens microRNA a partir de dades de seqüenciació massiva i comparació per homologia. Mitjançant una aproximació basada en grafs i un algoritme transductiu de Machine Learning, vam poder incrementar la quantitat d'informació disponible en l'entrenament del nostre model classificador, millorant així la seva capacitat predictiva respecte a la d'altres algoritmes prèviament publicats.
A més, vam demostrar l'aplicabilitat pràctica d'aquesta nova eina utilitzant dades de seqüenciació massiva de microRNAs en múscul de porcs Duroc. Un total de 47 microRNAs no inclosos en el catàleg porcí, i per tant l'existència dels quals es desconeixia, van ser detectats mitjançant el model implementat en eMIRNA, i d’aquests un total de 20 es van identificar en les mostres de múscul porcí.
L'eina eMIRNA, en definitiva, permet predir i identificar amb una elevada fiabilitat els gens microRNA de qualsevol espècie animal, així com caracteritzar funcionalment les seves dianes metabòliques, proporcionant l'oportunitat de millorar els catàlegs de gens microRNA en nombroses espècies amb anotacions genòmiques encara molt limitades. Aquesta tasca resulta indispensable per entendre en profunditat, per exemple, la funció dels microRNAs en la regulació de nombrosos caràcters productius d'interès per al sector ramader porcí.
Emilio Mármol Sánchez1 i Marcel Amills1,2
1Centre de Recerca Agrigenòmica.
2Universitat Autònoma de Barcelona.
Referències
Mármol-Sánchez E, Cirera S, Quintanilla R, Pla A, Amills M. (2020). Discovery and annotation of novel microRNAs in the porcine genome by using a semi-supervised transductive learning approach. Genomics 112: 2107-2118 doi: https://www.sciencedirect.com/science/article/pii/S0888754319304884?via%3Dihub