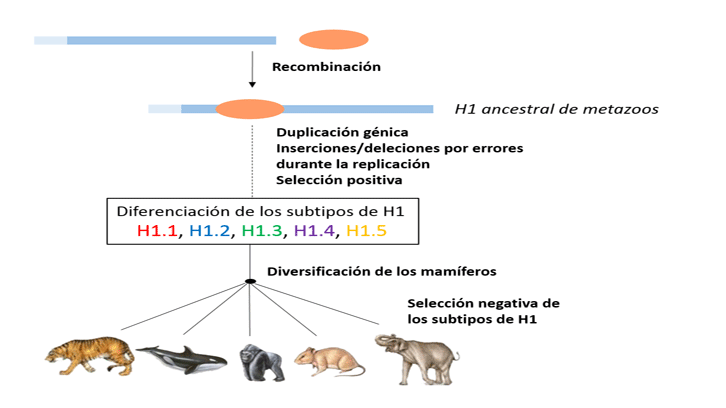
Historia evolutiva de la familia de histonas H1.1-H1.5 de mamíferos
En las células eucariotas la cromatina está formada por un complejo de DNA y proteínas, principalmente histonas, que permiten la compactación de todo el genoma dentro del pequeño volumen nuclear. El nucleosoma, la unidad fundamental de la cromatina, está formado por un octámero de las histonas H2A, H2B, H3 y H4, el DNA y la histona H1. La H1 participa en la regulación de la compactación de la cromatina y de la expresión génica.
La histona H1 tiene una estructura tripartita. El dominio central (G) se pliega establemente, mientras que los dominios N- y C-terminales están intrínsecamente desordenados, pero se pliegan al unirse al DNA. En los mamíferos hay 7 subtipos somáticos, H1.1-H1.5, H1.0 y H1x. Los subtipos H1.1-H1.5 se encuentran juntos en el genoma y aumentan su síntesis durante la fase S del ciclo celular. Su dominio central es prácticamente idéntico, mientras que los dominios terminales son más divergentes y tienen diferente longitud. A pesar de que todos los subtipos de H1 son capaces de unirse y compactar la cromatina, existen evidencias que sugieren su diferenciación funcional.
Nuestro grupo de investigación ha estudiado la evolución molecular de los subtipos H1.1-H1.5 en mamíferos. El análisis de un alineamiento de 130 secuencias de 27 especies diferentes mostró que el número y la posición de los residuos básicos están muy conservados en todos los subtipos, ya que la elevada carga positiva de la H1 es de vital importancia para su interacción con el DNA que está cargado negativamente. También se observa que muchas posiciones que son modificadas post-traduccionalmente están conservadas, especialmente los sitios de fosforilación.
Encontramos la presencia de un patrón de inserciones/deleciones (indels) en los dominios terminales entre los subtipos de una misma especie. Este patrón está conservado en todas las especies que hemos estudiado, lo que indica su aparición antes de la división de los órdenes de los mamíferos. Los dominios terminales están codificados por secuencias que contienen repeticiones, en las cuales son frecuentes errores de deslizamiento de hebra durante la replicación (slippage) que favorecen inserciones y deleciones cortas. Este mecanismo molecular explicaría la rápida evolución de los dominios terminales de los subtipos de H1. La conservación del patrón de indels en los subtipos de la H1 indica que este patrón está asociado a su diferenciación funcional.
El análisis con herramientas de evolución molecular ha mostrado que el subtipo H1.1 presenta evidencias de selección positiva o adaptativa en siete posiciones distribuidas en los 3 dominios estructurales. Los cambios encontrados en la H1.1 incluyen la disminución de la carga positiva, la disminución de la estabilidad del plegamiento del dominio globular y la posible modulación del plegamiento del dominio C-terminal. Por lo tanto, la selección positiva en la H1.1 puede asociarse a una disminución de su afinidad por la cromatina, lo que representaría una adaptación de este subtipo a su función específica, relacionada con procesos en que es necesario que la cromatina esté más relajada (replicación y espermatogénesis).
Detectamos posibles puntos de recombinación entre los dominios terminales y el dominio globular, que junto a otras evidencias descritas por otros grupos, sugiere que este proceso puede haber participado en la adquisición de la estructura tripartita de la H1. Integrando los resultados hemos elaborado un modelo de evolución de la familia multigénica de las histonas H1 (H1.1-H15) que corrobora la existencia de la diferenciación funcional de los subtipos.
Facultad de Biociéncias
Universitat Autònoma de Barcelona
Referencias
Ponte I, Romero D, Yero D, Suau P, Roque A. Complex Evolutionary History of the Mammalian Histone H1.1-H1.5 Gene Family. Mol Biol Evol. 2017;34(3):545-558. doi.org/10.1093/molbev/msw241